研究方向
| 表观遗传调控 |
 |
哺乳动物DNA甲基化发生在胞嘧啶第五位碳原子上,称为5-甲基胞嘧啶,是重要的表观遗传修饰,调控基因转录,基因组印记,表观遗传等多种生物学过程,在发育过程中起关键作用。哺乳动物基因组的DNA甲基化主要发生在CpG二核苷酸。其建立是由DNMT3A/3B在胚胎发育早期完成的,而甲基化模式的维持是有DNMT1和UHRF1实现的。TET蛋白能够氧化5-甲基胞嘧啶成为5-羟甲基胞嘧啶,5-醛基胞嘧啶,5-羧基胞嘧啶,在DNA去甲基化过程中起关键作用。我们课题组集中研究了上述蛋白质的催化,底物识别和酶活性调节的分子机制,并设计小分子调节剂用于研究,开展用于临床的药物开发。
|
DNA甲基化建立机制
|
| DNMT3A的自抑制及其被组蛋白H3激活的机制研究 |
|
 MOVIE MOVIE |
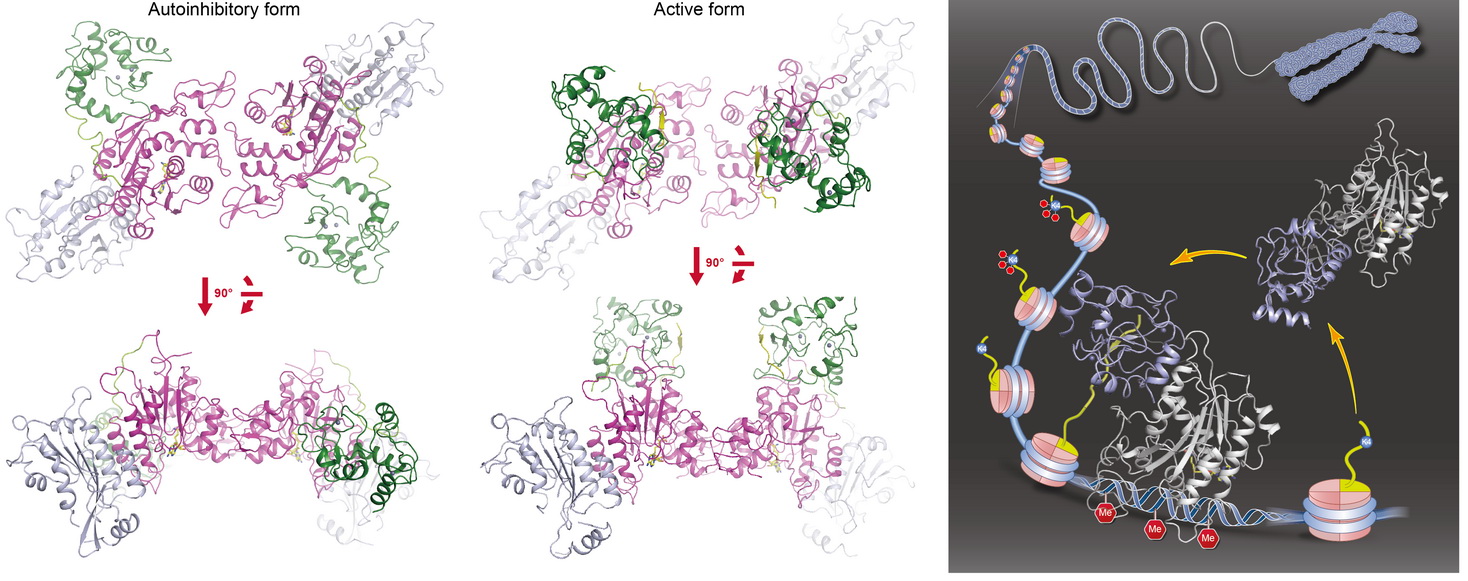
组蛋白和DNA甲基化存在相互调控。我们发现DNMT3A存在自我抑制状态,而这种抑制可以被组蛋白H3所解除。我们解析抑制态(左侧)和激活态(中间),两种状态的晶体结构,并开展生化研究 ( Nature,2015a)。发现DNMT3A的ADD结构域结合催化结构域(CD),通过抑制CD与DNA底物的结合抑制其活性。组蛋白H3结合ADD结构域,破坏ADD与CD的结合,使CD能够接触底物,进而激活DNMT3A的活性,由此提出DNMT3A自我抑制及其被组蛋白激活的的分子机制模型。这一结果提示DNA甲基化主要发生在有组蛋白H3存在的核小体附近,并很好的支持了以往基因组测序结果,即组蛋白H3K4me3和DNA甲基化负相关。 国际同行评价:F1000 |
DNA甲基化去除机制
|
| TET介导5-甲基胞嘧啶氧化的分子机制 |
|
 MOVIE MOVIE |
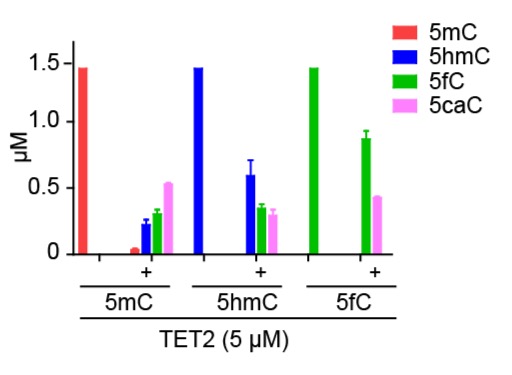
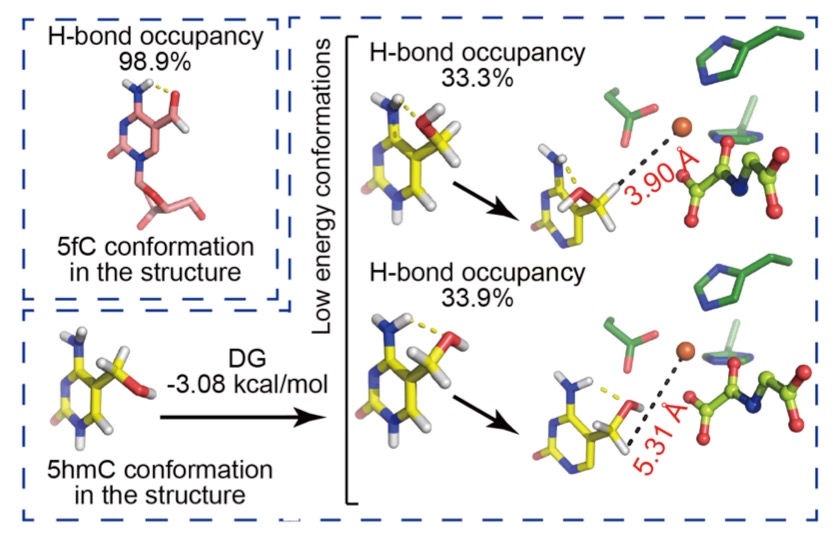
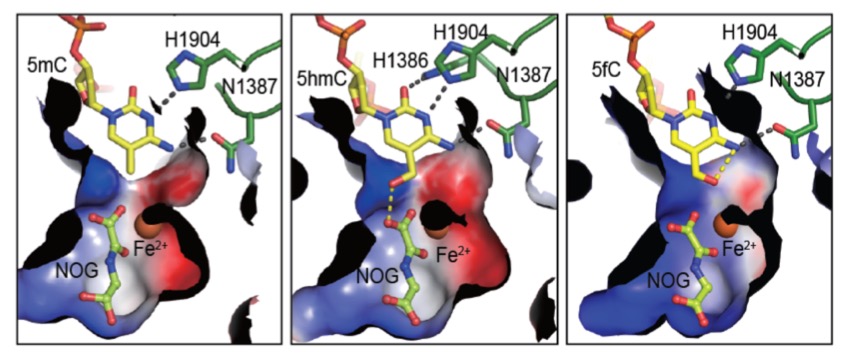
哺乳动物TET蛋白在受精卵表观遗传重编程、多能干细胞分化、骨髓造血等关键生命过程中扮演着至关重要的角色,其失活也与多种疾病,尤其是血液肿瘤的发生密切相关。TET 蛋白属于a-酮戊二酸(a-KG)和二价铁离子(Fe2+)依赖的双加氧酶,利用分子氧将5mC氧化为5-羟甲基胞嘧啶(5hmC),并继续催化5-hmC转化为5-甲酰胞嘧啶(5-fC)和5-羧基胞嘧啶(5-caC)。我们成功制备了TET2蛋白并解析了其与甲基化DNA的复合物结构 ( Cell, 2013)。TET2蛋白半胱氨酸富集结构域(Cys-Rich)“缠绕”着羧基端的催化结构域(DSBH),双链DNA位于催化结构域上方。结构分析和生化实验证明TET蛋白特异性识别CpG位点的DNA序列。 5mC通过碱基翻转的机制,被TET2从双链DNA中“撬开”,进而5mC翻出来并插入到催化口袋中,使5mC的甲基指向具有催化活性的二价铁离子(Fe2+)和a-酮戊二酸(a-KG),进而完成5mC的氧化过程。该研究为TET蛋白介导的DNA甲基化动态调控机制提供了新视角,高分辨率TET2-DNA复合物结构的解析,也为靶向TET蛋白激活剂和抑制剂的研究奠定了结构生物学基础。 国际同行评价: Faculty of 1000, Nature China, Preview in Cell |
DNA甲基化维持机制 |
| DNMT1稳定性受到USP7调节 |
DNMT1维持型DNA甲基转移酶,能够将DNA复制产生的半甲基化DNA,转化为全甲基化DNA,以维持基因组DNA甲基化的模式。USP7(去泛素化酶)结合并稳定DNMT1。我们解析了DNMT1-USP7复合物晶体结构,发现DNMT1的KG-linker(KG重复序列)结合到USP7的UBL1-2结构域的酸性表面,对复合物形成至关重要,细胞内实验发现KG-linker中(K1111等赖氨酸)的乙酰化,破坏DNMT1-USP7的结合导,使USP7不能够稳定DNMT1,导致DNMT1降解,进一步研究发现在肿瘤细胞和神经细胞发育过程中,乙酰化KG-Linker与整体DNMT1蛋白水平呈负相关( Nature Communications, 2015)。 |
 |
| |
| UHRF1识别组蛋白修饰的分子机制 |
 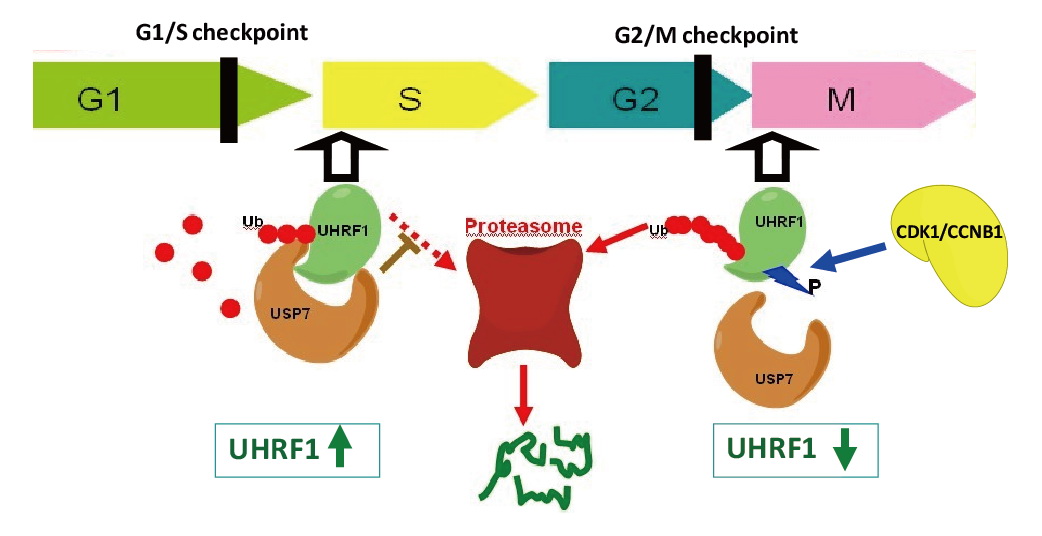 |
UHRF1 是联系DNA甲基化与组蛋白甲基化的关键蛋白。我们发现其PHD结构域特异性识别组蛋白H3的第二位精氨酸(H3R2)( Cell Res, 2021),其TTD结构域和PHD结构域共同作用,使UHRF1特异性识别组蛋白H3K9me3( JBC, 2013). 进一步研究发现USP7结合并稳定UHRF1,而UHRF1的磷酸化破坏二者结合,使UHRF1降解,揭示了UHRF1蛋白水平随细胞周期动态变化的机制( PNAS, 2012). |
 |
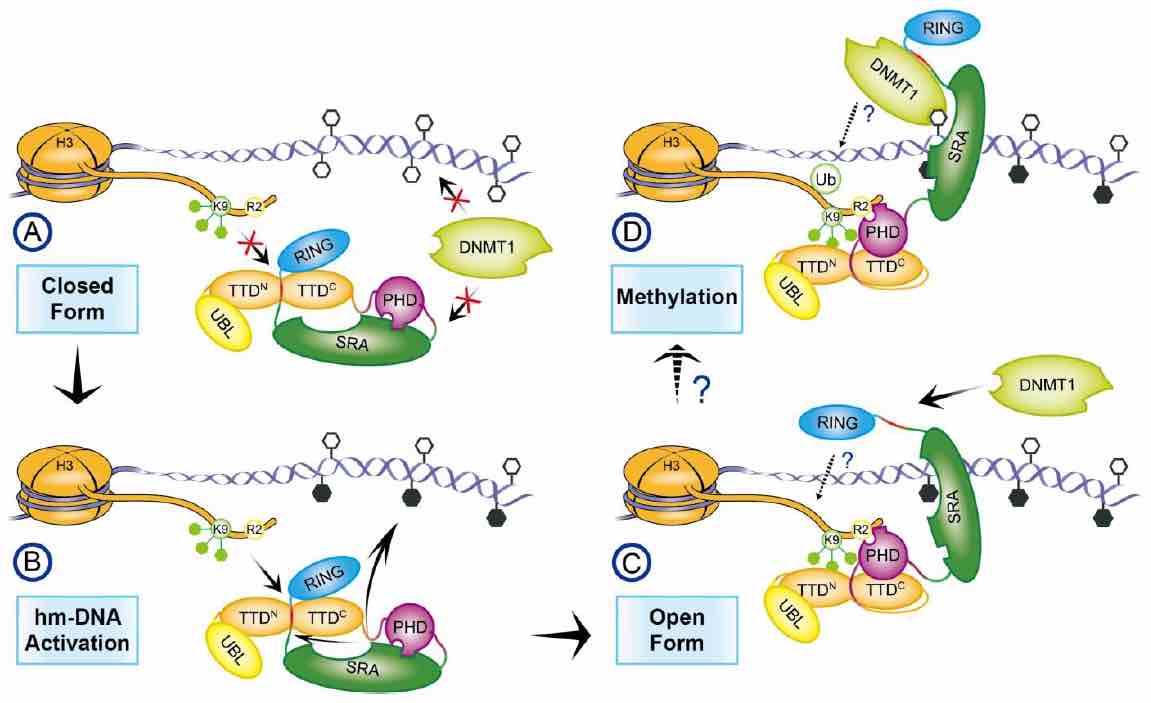 |
UHRF1的TTD结构域与Spacer序列结合,使UHRF1形成一种闭合构象,导致UHRF1对组蛋白 H3K9me3的结合强度很低,且无法招募DNMT1。UHRF1在结合hm-DNA后可由这种闭合构象转变为开放构象,使其恢复对组蛋白H3K9me3 的识别能力,并可与DNMT1相互作用。由此推断UHRF1对DNMT1的招募也受到自身构象变化的调控。该研究工作拓展了对DNA甲基化与组蛋白修饰之 间相互调节的深入理解,为进一步研究DNA甲基化动态调控奠定了基础( Nature Communications, 2016)。 |
组蛋白去甲基化酶的结构功能研究 |
|
|
 |
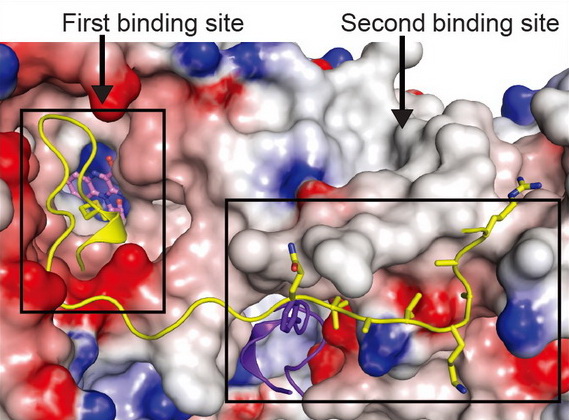
组蛋白去甲基化酶LSD2,是第一个被发现的组蛋白去甲基化酶LSD1的唯一同源物,与基因的转录延伸以及一些印记基因的甲基化相关。我们发现LSD2对H3(1-26氨基酸)的结合能力以及酶活性与H3(1-21氨基酸)相比,都比较高。解析了LSD2与H3(1-26)的复合物结构,发现LSD2与底物的结合除了酶活中心外,还有第二个底物识别位点,而LSD1不具有此部分序列,通过体内外结合以及酶活试验证明,第二个结合位点对于LSD2的酶活有重要的作用( Cell Res, 2013)。我们进一步发现NPAC蛋白可以显著提高LSD2的酶活性,解析了LSD2-NPAC-H3复合物的结构,发现NPAC结合LSD2并稳定其与底物H3的结合进而提高酶活性( Mol Cell, 2013, 国际同行评价:Nature China)。 |
|
进一步研究发现LSD2具有泛素连接酶活性,可以直接泛素化OGT并促进其降解。LSD2通过其E3连接酶活性抑制癌细胞生长,这一效果不依赖于其组蛋白去甲基化酶活性。因此,LSD2通过组蛋白去甲基化酶和E3连接酶活性调节不同的基因和细胞功能( Mol Cell,2015, 国际同行评价:Science Signaling)。 |
|
|
|